0. AlphaFold2 之后:蛋白结构预测对小分子药物发现的影响
1. 引言:AlphaFold2时代的蛋白结构预测革新及其对小分子药物发现的深远意义
本综述旨在系统性地分析AlphaFold2 (AF2) 在小分子药物发现全流程中的具体应用、现有局限性及其深层原因,并提出跨学科的未来解决方案,从而超越简单的成果罗列,为该领域研究提供更全面的指导。本报告将首先概述AlphaFold2如何改变了传统的蛋白结构预测范式,突出其在预测精度、效率和可及性上的飞跃,并解释这种提升对后续研究和应用(尤其是药物发现)的深远意义。通过对比AlphaFold2与传统方法在解决蛋白质折叠问题上的优势,本综述将强调其作为一种通用工具的潜力,并简要概述小分子药物发现的基本流程,进而分析传统药物发现中与蛋白结构获取相关的痛点,预设AlphaFold2如何通过提供“更广泛、更快速”的结构信息来直接或间接地缓解这些瓶颈。
在AlphaFold2出现之前,蛋白质三维结构的解析主要依赖于X射线晶体学、核磁共振(NMR)和冷冻电镜(Cryo-EM)等实验技术。这些方法在解析精度上具有优势,但普遍面临耗时冗长、成本高昂以及成功率有限等固有挑战,尤其对于膜蛋白等难以结晶的靶点,其局限性更为突出。这些挑战严重阻碍了蛋白质结构信息的可及性,进而限制了基于结构的药物设计(SBDD)的效率和广度。
AlphaFold2的问世标志着蛋白质结构预测领域的范式突破,其核心在于利用深度学习技术,特别是注意力机制和进化信息,从氨基酸序列直接预测蛋白质的三维结构,并达到接近实验测定的精度。在第14届关键评估蛋白质结构预测(CASP)竞赛中,AlphaFold2展现出超越以往所有方法的预测精度,其GDT_TS(全局距离测试_总分)中位数通常在90分以上,这意味着其预测结果在很大程度上与实验解析结构高度吻合,从而使计算生物学进入了一个新时代。
AlphaFold2通过其深度学习架构,显著克服了传统实验方法在效率、成本和适用性上的局限性,实现了预测精度、效率和可及性的飞跃。这种高精度、高效率和高可及性的特点,使其能够加速对未知或难以结晶蛋白质结构的解析,为药物发现提供了前所未有的结构信息可及性,开启了计算生物学的新时代。AlphaFold2的出现不仅为计算生物学带来了革命性的进展,也为药物发现领域打开了新的大门,加速了基于结构的药物设计。AlphaFold2对传统湿实验依赖的降低以及对早期药物发现阶段的加速效应,构成了其在概念和实践层面的具体影响。其作为一种通用工具的潜力,在于能够为广泛的蛋白质靶点提供高精度结构,有效降低了“假阳性/假阴性”结构预测的风险,进而提高药物发现的成功率。此外,AlphaFold2还促使研究范式从传统的“经验驱动”转变为“数据驱动+计算引导”,显著提升了科研效率和创新能力。
尽管AlphaFold2取得了显著成就,但其预测主要侧重于单一稳定构象,对于蛋白质的动态性、构象柔性以及配体诱导的构象变化仍存在局限性。这对于药物发现,特别是构象选择性药物设计构成了挑战,因为理解蛋白质在结合过程中的动态变化至关重要。未来研究需进一步探索如何整合蛋白质动态信息,以充分发挥AlphaFold2在药物发现中的潜力。
小分子药物发现是一个高度复杂、资源密集且风险巨大的过程,其传统流程涵盖靶点识别与验证、先导化合物筛选、先导化合物优化、临床前研究以及临床试验等多个关键阶段。然而,这一漫长且昂贵的历程面临诸多挑战,包括居高不下的失败率(临床试验失败率高达90%)、漫长的研发周期(平均10-15年)以及巨额的研发成本(平均20-30亿美元)。在传统药物发现过程中,获取精确的靶点蛋白结构信息是一个核心瓶颈。许多重要的疾病靶点,尤其是膜蛋白,由于其固有的复杂性和在细胞膜中的嵌入特性,极难通过X射线晶体学或冷冻电镜(Cryo-EM)等实验方法获得高分辨率结构。这种结构信息的缺失严重阻碍了基于结构的药物设计(SBDD)的应用,导致虚拟筛选效率低下,药物设计和优化过程受阻,从而影响药物的特异性和疗效。
除了静态结构获取的挑战,蛋白质的动态构象变化在药物结合过程中扮演着关键角色。靶点蛋白在与配体结合时会经历复杂的构象重排,这些动态性对于理解药物作用机制和实现构象选择性药物设计至关重要,特别是对于靶向特定构象的抑制剂(如II型抑制剂)。然而,传统的结构获取方法以及AlphaFold2的静态预测能力难以充分捕捉这些动态性,限制了构象选择性药物设计的发展,是当前药物发现领域的一大瓶颈。
AlphaFold2的出现,凭借其在蛋白质结构预测方面的显著进展,有望缓解上述结构信息瓶颈。通过提供更广泛、更快速的准确蛋白质结构信息,AlphaFold2能够赋能药物发现的各个环节,尤其是在面对传统实验方法难以解析的靶点蛋白时,为基于结构的药物设计提供了前所未有的机遇,从而加速药物发现流程,例如在肝癌新药设计中的应用已初步展现其潜力。尽管AlphaFold2主要预测静态结构,对蛋白质动态性的捕获仍有局限,但其为后续章节讨论如何克服这些挑战,并通过结合先进计算工具进一步深化构象选择性药物设计奠定了基础。
1.1 蛋白结构预测的里程碑式进展:从传统挑战到AlphaFold2的范式突破
在AlphaFold2出现之前,蛋白质三维结构的解析主要依赖于X射线晶体学、核磁共振(NMR)和冷冻电镜(Cryo-EM)等实验技术 。尽管这些方法在解析精度上具有优势,但其普遍面临耗时冗长、成本高昂以及成功率有限等固有挑战,尤其对于膜蛋白等难以结晶的靶点,其局限性更为突出 。这些挑战严重阻碍了蛋白质结构信息的可及性,进而限制了基于结构的药物设计(SBDD)的效率和广度。
AlphaFold2的问世标志着蛋白质结构预测领域的范式突破,其核心在于利用深度学习技术,尤其是注意力机制和进化信息,从氨基酸序列直接预测蛋白质的三维结构,并达到接近实验测定的精度 。在第14届关键评估蛋白质结构预测(CASP)竞赛中,AlphaFold2展现出超越以往所有方法的预测精度,其GDT_TS(全局距离测试_总分)中位数通常在90分以上,这意味着其预测结果在很大程度上与实验解析结构高度吻合,从而使计算生物学进入了一个新时代 。
AlphaFold2通过其深度学习架构,显著克服了传统实验方法在效率、成本和适用性上的局限性,实现了预测精度、效率和可及性的飞跃 。这种高精度、高效率和高可及性的特点,使其能够加速对未知或难以结晶蛋白质结构的解析,为药物发现提供了前所未有的结构信息可及性,开启了计算生物学的新时代 。AlphaFold2的出现不仅为计算生物学带来了革命性的进展,也为药物发现领域打开了新的大门,加速了基于结构的药物设计 。
AlphaFold2对传统湿实验依赖的降低以及对早期药物发现阶段的加速效应,构成了其在概念和实践层面的具体影响。其作为一种通用工具的潜力,在于能够为广泛的蛋白质靶点提供高精度结构,有效降低了“假阳性/假阴性”结构预测的风险,进而提高药物发现的成功率。此外,AlphaFold2还促使研究范式从传统的“经验驱动”转变为“数据驱动+计算引导”,显著提升了科研效率和创新能力 。
尽管AlphaFold2取得了显著成就,但其预测主要侧重于单一稳定构象,对于蛋白质的动态性、构象柔性以及配体诱导的构象变化仍存在局限性 。这对于药物发现,特别是构象选择性药物设计构成了挑战,因为理解蛋白质在结合过程中的动态变化至关重要。未来研究需进一步探索如何整合蛋白质动态信息,以充分发挥AlphaFold2在药物发现中的潜力。
1.2 小分子药物发现的背景与结构信息瓶颈
小分子药物发现是一个高度复杂、资源密集且风险巨大的过程,其传统流程涵盖靶点识别与验证、先导化合物筛选、先导化合物优化、临床前研究以及临床试验等多个关键阶段。然而,这一漫长且昂贵的历程面临诸多挑战,包括居高不下的失败率(临床试验失败率高达90%)、漫长的研发周期(平均10-15年)以及巨额的研发成本(平均20-30亿美元)。
在传统药物发现过程中,获取精确的靶点蛋白结构信息是一个核心瓶颈。许多重要的疾病靶点,尤其是膜蛋白,由于其固有的复杂性和在细胞膜中的嵌入特性,极难通过X射线晶体学或冷冻电镜(Cryo-EM)等实验方法获得高分辨率结构。这种结构信息的缺失严重阻碍了基于结构的药物设计(SBDD)的应用,导致虚拟筛选效率低下,药物设计和优化过程受阻,从而影响药物的特异性和疗效。
除了静态结构获取的挑战,蛋白质的动态构象变化在药物结合过程中扮演着关键角色。靶点蛋白在与配体结合时会经历复杂的构象重排,这些动态性对于理解药物作用机制和实现构象选择性药物设计至关重要,特别是对于靶向特定构象的抑制剂(如II型抑制剂)。然而,传统的结构获取方法以及AlphaFold2的静态预测能力难以充分捕捉这些动态性,限制了构象选择性药物设计的发展,是当前药物发现领域的一大瓶颈。
AlphaFold2的出现,凭借其在蛋白质结构预测方面的显著进展,有望缓解上述结构信息瓶颈。通过提供更广泛、更快速的准确蛋白质结构信息,AlphaFold2能够赋能药物发现的各个环节,尤其是在面对传统实验方法难以解析的靶点蛋白时,为基于结构的药物设计提供了前所未有的机遇,从而加速药物发现流程,例如在肝癌新药设计中的应用已初步展现其潜力。尽管AlphaFold2主要预测静态结构,对蛋白质动态性的捕获仍有局限,但其为后续章节讨论如何克服这些挑战,并通过结合先进计算工具进一步深化构象选择性药物设计奠定了基础。
2. AlphaFold2对结构引导药物发现(SBDD)流程的深度赋能与AI协同
AlphaFold2的出现标志着蛋白质结构预测领域的一项革命性突破,它显著缓解了传统药物发现过程中长期存在的瓶颈,尤其在加速结构引导药物发现(SBDD)的各个关键环节中展现出深远的赋能作用 。本章旨在深入剖析AlphaFold2如何通过提供高精度蛋白质结构,具体提升SBDD的效率和智能化水平。
AlphaFold2的核心贡献在于其通过预测蛋白质结构,极大地加速了靶点结构获取、显著提高了小分子设计与优化的效率,并进一步加速了药物筛选与重利用的进程 。这些突破不仅限于单一环节的改进,更在于其与人工智能(AI)技术的深度协同,共同推动了药物发现流程的整体智能化和自动化 。通过整合AlphaFold2生成的高质量结构信息与AI在数据分析、模式识别和预测方面的强大能力,研究人员能够更有效地识别潜在药物靶点、设计和优化先导化合物,并进行高通量药物筛选,从而显著缩短了从基础研究到临床应用的整体药物研发周期 。本章将系统性地阐述AlphaFold2如何通过上述机制赋能SBDD,并强调AI在其中所扮演的关键协同角色,以实现药物发现全流程的智能化转型。
2.1 加速靶点结构获取与识别
AlphaFold2的问世极大地加速了药物靶点结构的获取,尤其对于那些传统实验方法难以解析的蛋白质,如膜蛋白或柔性蛋白,其效用尤为显著。在缺乏实验结构信息的情况下,AlphaFold2能够提供高置信度的结构模型供药物设计使用,这为结构生物学和药物发现领域带来了变革性的影响。
AlphaFold2相较于传统的实验结构解析方法(如X射线晶体学或冷冻电镜)在效率和成本效益方面展现出显著优势。通过预测蛋白质结构,AlphaFold2显著降低了前期结构获取的门槛和成本,使得研究人员能够更快地进入药物设计的后续阶段,从而加速了潜在药物靶点的识别和验证过程。这种高精度预测能力对于未知或难以通过传统方法解析的蛋白质结构尤为关键,为靶点发现和验证提供了新的途径。AlphaFold2所提供的预测结构可以作为虚拟筛选、分子对接以及其他基于结构药物设计的起点,极大地克服了传统基于结构药物设计(SBDD)在靶点结构获取方面的瓶颈。
尽管AlphaFold2在提供高精度靶点结构方面表现卓越,但在实际应用中仍面临特定挑战。其中一个主要挑战是对预测结构置信度的评估。虽然AlphaFold2提供了置信度指标(如pLDDT),但其在所有情况下的可靠性和对下游药物设计过程的直接影响仍需进一步验证。此外,对于某些动态或柔性区域的蛋白质,AlphaFold2的单一结构预测可能无法完全捕捉其构象多样性,而构象多样性在药物结合和功能调控中至关重要。未来的研究方向可能包括结合多构象采样方法或集成分子动力学模拟,以更好地理解和利用AlphaFold2预测结构的动态特性。
2.2 小分子设计与优化的效率提升
AlphaFold2预测的蛋白质结构已在基于结构的虚拟筛选(SBVS)和药物发现中展现出显著的应用潜力,直接提升了小分子设计与优化的效率。这些高精度结构能够精确识别配体结合位点,从而显著提高分子对接的准确性,并加速命中化合物的发现过程。
AlphaFold2预测结构直接集成到SBVS流程中,通过提供详尽的蛋白质结构信息,引导配体发现,并改进小分子配体的设计和优化。例如,在对接研究中,AlphaFold2结构能为化合物与靶点蛋白的相互作用提供精确的几何构象信息,从而指导药物化学家进行更为合理的分子修饰和优化。这种结构信息的提供,使得研究人员能够更准确地预测配体-蛋白的结合模式,从而有效地促进命中化合物的发现。
在虚拟筛选和药物优化阶段,AlphaFold2的贡献尤为突出。通过提供精确的靶点结构信息,AlphaFold2使得药物设计者能够更深入地理解药物与靶点之间的相互作用,进而优化化合物的结合亲和力和选择性。尽管AlphaFold2本身不直接进行小分子设计,但其所提供的准确蛋白结构是促进虚拟筛选和基于结构药物设计(SBDD)的基础,有助于加速配体设计和优化过程。此外,结合人工智能(AI)模型,AlphaFold预测的结构已被用于指导AI模型进行小分子配体设计和优化,进一步提升了药物设计和优化的效率,例如在肝癌新型药物设计中已取得进展。这种集成应用体现了“加速药物发现周期”的核心理念,即通过高效的结构预测加速后续的药物设计和优化环节。
然而,在这一阶段也面临一些挑战。其中一个主要问题是结合亲和力预测的准确性。尽管AlphaFold2提供了高精度的蛋白质结构,但精确预测小分子与这些结构之间的结合亲和力仍是一个复杂且需要进一步优化的领域。这涉及到更深层次的分子动力学模拟和自由能计算,以充分捕捉配体结合过程中的动态变化和溶剂效应。未来的研究需要重点解决这些挑战,以充分发挥AlphaFold2在小分子药物发现中的潜力。
2.3 加速药物筛选与重利用
AlphaFold2在高精度蛋白质结构预测方面的突破,显著加速了药物筛选和药物重利用的进程。该技术通过提供高置信度的靶点结构,极大地拓展了可筛选蛋白质的范围,并提升了虚拟筛选的效率。
首先,AlphaFold2间接赋能了大规模的药物筛选,通过提供精确的蛋白质结构信息,为分子对接和虚拟筛选提供了更有效的起点。研究人员能够利用AlphaFold2预测的靶点结构进行高通量虚拟筛选,从而快速识别潜在的配体并预测药物-靶点之间的相互作用模式。这种基于结构的方法能够显著降低实验筛选的成本和时间,加速新药的发现过程。
其次,在药物重利用方面,AlphaFold2展现出巨大的潜力。通过将已知药物与预测的靶点结构进行分子对接,研究人员可以快速评估现有药物的潜在新适应症,从而为药物重利用提供结构基础。这不仅有助于发现药物的新用途,还能够缩短新药上市时间并降低研发成本。尽管现有研究尚未提供具体案例,但AlphaFold2通过提供准确的结构信息,已间接证明其在赋能药物重利用方面的能力。
然而,尽管AlphaFold2带来了显著优势,其在药物重利用中仍面临挑战。例如,AlphaFold2主要关注单一蛋白质结构预测,而药物与靶点结合的复杂性,特别是涉及蛋白质构象变化和多蛋白复合物的情况,仍需进一步探索。此外,脱靶效应的预测也是药物重利用中的关键难题,AlphaFold2目前尚未能直接解决这一问题。未来研究应着重于将AlphaFold2与其他计算方法结合,以更全面地预测药物-靶点相互作用、评估脱靶效应,并解决蛋白质动态和多构象状态下的结合模式复杂性问题。
2.4 AlphaFold2与AI在药物设计中的深度协同与案例分析
AlphaFold2作为人工智能(AI)在蛋白质结构预测领域的典范应用,其通过深度学习驱动实现了结构预测的范式变革,本身即为AI驱动的工具。其高精度的结构信息为AI驱动的药物发现平台提供了关键的结构输入,从而与AI在数据分析、模式识别和预测方面的能力互补,共同促进了药物发现流程的自动化和智能化。这种深度协同显著提升了药物发现的效率,加速了化合物的生成、筛选与优化进程,从而缩短了整体药物研发周期。
在药物设计中,AlphaFold2预测的蛋白质结构能够作为机器学习模型(如亲和力预测模型)的输入,或者作为生成式AI模型(如扩散模型)的输入,用于生成具有特定结合模式的配体。这种整合使得AI能够更有效地进行分子生成、虚拟筛选和性质预测。例如,一项研究成功将AlphaFold与AI技术深度融合,用于设计新型肝癌药物,并验证了其在实验中的积极疗效,为AlphaFold与AI在实际药物发现中的协同效应提供了具体案例支持。此外,AlphaFold2-RAVE的开发通过整合AlphaFold2的静态结构预测能力与分子动力学模拟及先进采样技术,旨在识别和利用蛋白质的动态构象,从而促进构象选择性药物设计,为结构生物学辅助药物设计(SBDD)提供了更丰富的结构信息,以提升药物发现的效率和成功率。
尽管AlphaFold2与AI的深度协同展现出巨大潜力,当前仍存在一些局限性。首先,在模型集成方面,如何有效地将AlphaFold2的结构预测与各种下游AI模型(如生成模型、亲和力预测模型)无缝整合,并实现端到端的优化,仍是需要深入研究的领域。其次,数据标准和可解释性问题依然突出。在AI驱动的药物发现中,数据质量和标准化对模型性能至关重要,而许多研究并未详细披露AI模型的具体算法、架构和训练细节,限制了其在其他靶点或疾病中推广应用的潜力。此外,AI模型的“黑箱”特性使得其决策过程难以解释,这在需要高安全性和可追溯性的药物研发领域构成挑战。
展望未来,AI在整合AlphaFold2后具有在生成式设计、强化学习优化等方面的巨大潜力。通过利用生成式AI模型设计具有特定药理活性的新型分子,或运用强化学习优化分子设计过程以最大化药效并最小化毒副作用,将进一步加速药物研发进程。例如,在多靶点药物设计领域,AlphaFold2能够为多个靶点提供精确的结构信息,为AI模型开发具有多重药理活性的化合物提供基础。随着AI技术和计算能力的不断发展,AlphaFold2与AI的协同作用将持续推动药物发现向更高效率、更智能化、更精准化的方向发展。
3. AlphaFold2在药物发现中的挑战、局限性与应对策略
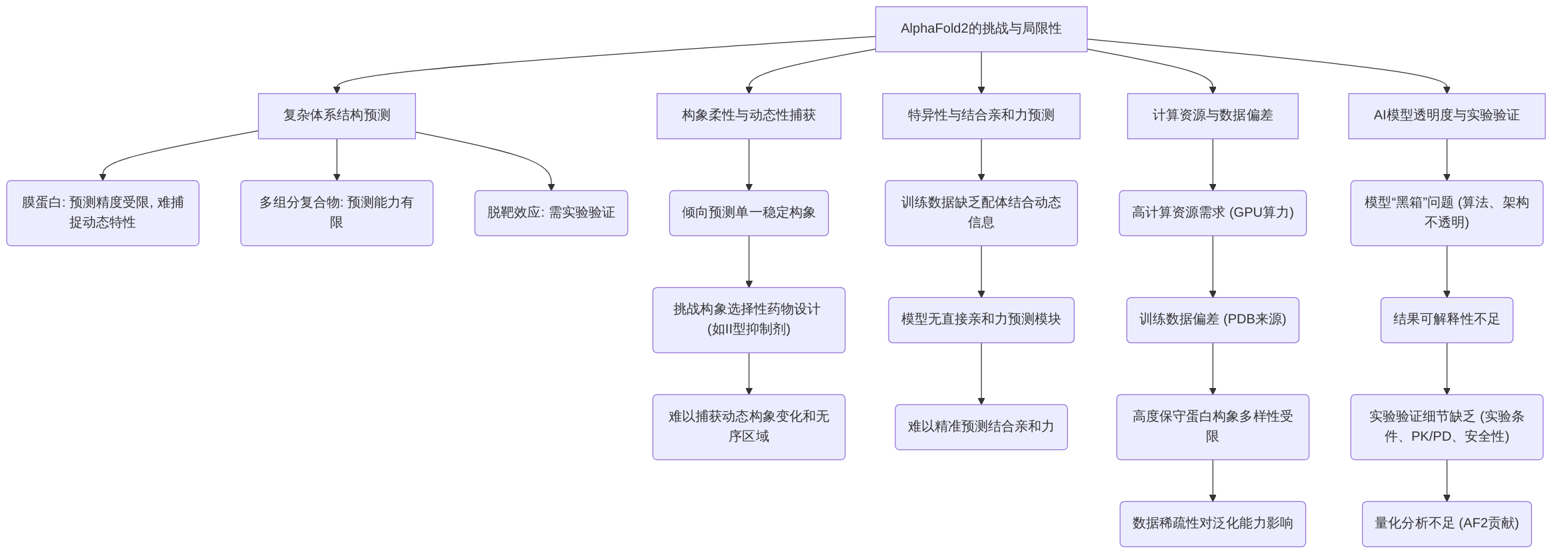
AlphaFold2的问世标志着蛋白质结构预测领域的一项里程碑式突破,极大地加速了结构生物学和药物发现的进程。然而,尽管其在预测可溶性蛋白质结构方面表现出前所未有的精度,AlphaFold2在赋能药物发现过程中仍面临一系列深层挑战和固有局限性。这些挑战不仅源于蛋白质自身复杂的多样性,也与模型训练数据、计算资源需求以及AI模型的可解释性等因素密切相关。本章旨在深入分析AlphaFold2在应用于药物发现时所遭遇的瓶颈,并系统性地探讨其根本原因以及当前及未来可能的应对策略。
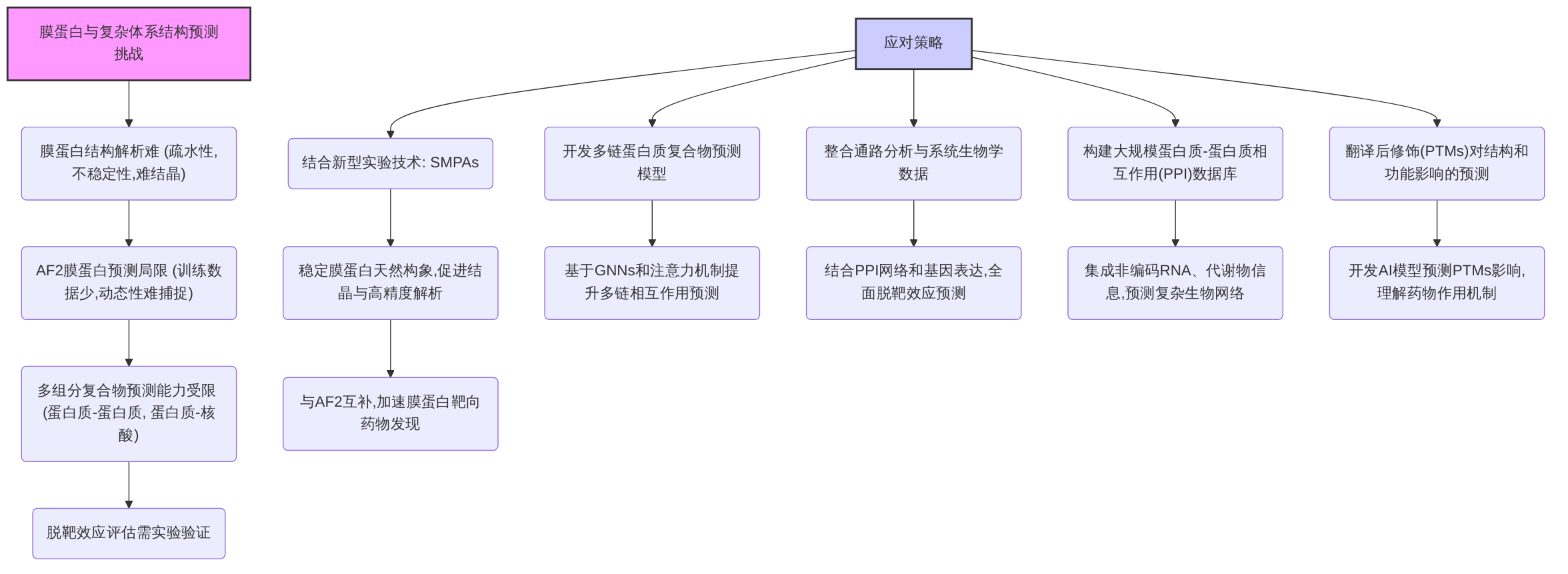
首先,AlphaFold2在处理复杂蛋白质体系时面临挑战,尤其是在膜蛋白和多组分复合物的结构预测方面。膜蛋白作为关键药物靶点,其结构解析的难度一直是药物发现领域的痛点,AlphaFold2虽然在此领域展现出一定潜力,但仍受限于训练数据中膜蛋白的稀缺性,导致预测精度不如实验方法,且难以准确捕捉其在脂质环境中的动态特性。此外,AlphaFold2主要以单体蛋白质为训练数据,其在预测蛋白质-蛋白质复合物、蛋白质-核酸复合物以及包含非标准氨基酸或修饰的蛋白质结构时,能力存在局限性,并且在评估脱靶效应时仍需依赖于实验验证和进一步的计算方法。
其次,AlphaFold2在捕获蛋白质构象柔性与动态性方面存在固有局限性,其主要倾向于预测单一的、最低能量的稳定构象。这对于构象选择性药物设计构成挑战,特别是对于需要靶向特定非活性构象或诱导构象变化的药物,如II型抑制剂,AlphaFold2直接生成的静态结构通常无法成功对接此类抑制剂,导致较高的假阴性率。蛋白质在生理条件下并非静态,其动态构象变化对于药物结合至关重要,AlphaFold2的静态预测能力限制了其在捕获这些动态过程以及无序区域构象方面的效用。
再者,AlphaFold2在特异性配体结合位点识别和结合亲和力预测方面仍存在固有局限性,这直接影响了药物发现过程中的效率与成功率。这些局限性主要源于AlphaFold2的训练数据以单体蛋白质结构为主,缺乏配体结合状态下的动态信息和复合物结构信息,且模型本身不包含直接的结合亲和力预测模块。
此外,AlphaFold2的大规模应用,特别是在药物发现研究中,面临着巨大的计算资源需求和数据偏差挑战。AlphaFold2的运行需要高性能计算集群和大量的图形处理器(GPU)算力,这对于中小型研究机构构成了显著的障碍。其预测性能还受到训练数据(主要来源于PDB数据库)潜在偏差的影响,导致模型在处理特定靶点或蛋白家族(如膜蛋白、柔性区域)时,尤其是在处理高度保守蛋白质时,生成多样化构象的能力受限。
最后,当前研究中普遍存在的AI模型透明度、可解释性以及实验验证不足的问题,严重限制了这些技术的普适性、可信度及其在临床转化中的应用。许多研究未能详细阐述所用AI模型的具体算法、架构和训练细节,形成了“黑箱”问题,阻碍了研究人员深入理解AI模型如何做出分子设计决策,从而削弱了其在不同靶点或疾病中推广应用的潜力,并使得模型的调试和改进变得困难。同时,在提及AI设计的分子在实验中显示出积极疗效时,普遍缺乏关于实验条件、细胞系/动物模型选择依据、关键药效学/药代动力学参数以及药物安全性评估的详细信息,导致结果的外部效度和可重复性受到影响。
综上所述,尽管AlphaFold2为药物发现带来了革命性的变革,但在膜蛋白与复杂体系预测、构象柔性、结合亲和力预测、计算与数据,以及模型可解释性与实验验证等多个维度,其仍面临显著的挑战与局限。本章将对这些瓶颈进行批判性评估,并提出前瞻性的解决方案,以期最大化AlphaFold2在药物发现中的潜力。
3.1 膜蛋白与复杂体系结构预测的挑战与应对
膜蛋白在药物发现中占据核心地位,约60%的已上市药物靶向膜蛋白,凸显了其作为关键药物靶点的战略重要性。然而,膜蛋白的结构解析是药物发现领域长期存在的重大挑战。传统实验方法,如X射线晶体学,因膜蛋白的疏水性、在水溶液中结构的不稳定性以及难以从细胞膜中提取并结晶等固有特性,难以获得其高分辨率结构。尽管冷冻电镜(Cryo-EM)为膜蛋白结构解析带来了突破,但其依然耗时耗力。
AlphaFold2的问世为膜蛋白结构预测带来了新的机遇。尽管AlphaFold2在可溶性蛋白结构预测方面表现卓越,但在处理膜蛋白时仍存在一定局限性,其预测精度可能不如实验方法,且难以准确捕捉膜蛋白在脂质环境中的动态特性。这主要是由于其训练数据集中膜蛋白的比例相对较低,从而影响了预测精度。尽管如此,AlphaFold2在膜蛋白结构预测方面已显示出优于其他同源建模方法的潜力。例如,AlphaFold2预测的G蛋白偶联受体(GPCRs)结构在跨膜螺旋区域与实验结构高度一致,均方根偏差(RMSD)可达到小于1Å的精度。然而,对于膜蛋白的胞内或胞外域,预测精度可能有所下降。
为应对这些挑战,EPFL的科学家开发了一种新型实验技术:稳定膜蛋白类似物(SMPAs)。SMPAs通过将脂质双层膜的天然构象用合成聚合物稳定化,从而形成一种模拟膜环境的结构,使膜蛋白能够在其中保持其天然构象并进行结晶。这项技术能够稳定膜蛋白并促进其结构解析,从而为结构引导药物设计提供精确的膜蛋白结构信息。SMPAs技术的引入与AlphaFold2的预测能力形成互补,可以在AlphaFold2预测精度有限的膜蛋白领域提供关键的实验验证和高质量数据,共同加速膜蛋白靶向药物的发现,尤其在结晶GPCRs方面已展现出潜力,为神经退行性疾病和癌症等疾病的治疗提供了新的靶点识别和药物设计途径。
除了膜蛋白结构预测的挑战,AlphaFold2在药物发现中的其他潜在局限性亦需关注。现有研究未能充分讨论AlphaFold2在预测蛋白质-配体相互作用复杂性、多聚体或大分子复合物结构、以及脱靶效应评估方面的能力。AlphaFold2主要以单体蛋白质为训练数据,因此在处理多组分复合物,如蛋白质-蛋白质复合物、蛋白质-核酸复合物以及含有非标准氨基酸或修饰的蛋白质结构时,其预测能力存在局限性,并且在评估脱靶效应时仍需依赖于实验验证和进一步的计算方法。
针对这些复杂体系在药物发现中日益重要性,未来研究需着重关注以下方向:
- 多链蛋白质复合物预测模型:开发基于图神经网络(GNNs)和注意力机制的新架构,使其能够同时处理多个蛋白质链之间的相互作用,从而提高蛋白质-蛋白质复合物及其他大分子复合物的预测精度。
- 整合通路分析与系统生物学数据:利用AlphaFold2预测的结构信息,结合蛋白质相互作用网络和基因表达数据进行通路分析和系统生物学研究,以实现更全面的脱靶效应预测和多靶点药物设计。
- 构建大规模蛋白质-蛋白质相互作用(PPI)数据库:集成非编码RNA、代谢物等信息,训练能够预测复杂生物网络中药物作用的模型,从而更准确地评估药物的体内行为和潜在副作用。
- 翻译后修饰(PTMs)对结构和功能影响的预测:开发能够预测翻译后修饰对蛋白结构和功能影响的AI模型,因为PTMs在细胞信号传导和疾病发生发展中扮演关键角色,对药物作用机制具有重要影响。
综合来看,AlphaFold2在蛋白质结构预测领域取得了显著进展,但其在膜蛋白和复杂体系预测方面仍面临挑战。通过与新兴实验技术如SMPAs的结合,并积极开发针对多链复合物、通路分析及PTMs预测的新模型,将能够弥补AlphaFold2的现有局限性,从而更全面地赋能小分子药物的发现与开发。
3.2 蛋白质构象柔性与动态性捕获的挑战与增强策略
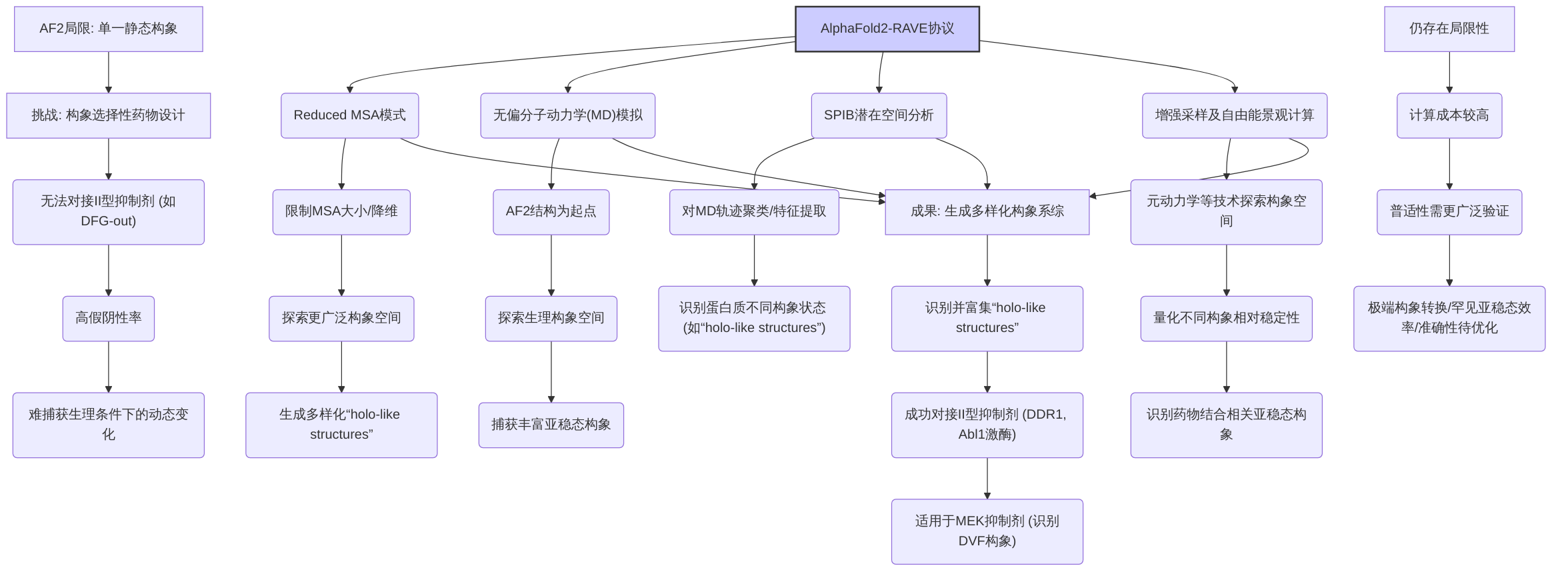
AlphaFold2在蛋白质结构预测领域取得了显著进展,但其在捕获蛋白质构象柔性与动态性方面存在固有局限性,主要倾向于预测单一的、最低能量的稳定构象。这种局限性对构象选择性药物设计构成挑战,特别是对于需要靶向特定非活性构象或诱导构象变化的药物,如II型抑制剂(例如针对DFG-out构象的激酶抑制剂)。AlphaFold2直接生成的静态结构通常无法成功对接此类抑制剂,导致较高的假阴性率。蛋白质在生理条件下并非静态,其动态构象变化(如变构效应和构象转换)对于药物结合至关重要,AlphaFold2的静态预测能力限制了其在捕获这些动态过程以及无序区域构象方面的效用。
为解决AlphaFold2在构象柔性和动态性预测上的不足,研究人员开发了AlphaFold2-RAVE(Reduced AlphaFold2 Variational Ensemble 或 Reconstructive AF2 Variational Ensemble 或 Re-sampled AlphaFold with Virtual Ensemble)协议。该协议旨在利用AlphaFold2预测的静态结构作为起点,通过整合多种计算方法来捕获蛋白质的动态构象系综,从而实现对蛋白质动态构象的精确预测。
AlphaFold2-RAVE协议的核心技术细节和创新点包括:
- Reduced MSA模式:该模式通过限制多序列比对(MSA)的大小或进行子采样/降维,减少对已知同源序列的依赖,使AlphaFold2不再提供足够信息以预测单一精确结构,而是引导模型探索更广泛的构象空间,生成更多样化的“holo-like structures”。这些构象可能与配体结合状态下的构象相似,从而克服AlphaFold2默认模式下构象多样性不足的问题。
- 无偏分子动力学(MD)模拟:利用AlphaFold2生成的结构作为初始点,进行长时间的无偏MD模拟,以探索蛋白质在生理条件下的构象空间,捕获更丰富的亚稳态构象,弥补AlphaFold2在动态性预测上的不足。
- SPIB(State Predictive Information Bottleneck)潜在空间分析:这是一种机器学习方法,用于对MD轨迹中的构象进行聚类和特征提取,将其投影到低维空间,从而识别和区分蛋白质的不同构象状态,尤其关注与功能相关的亚稳态构象,例如“holo-like structures”。SPIB有助于理解蛋白质在不同构象状态下的能量景观并量化构象之间的转移概率。
- 增强采样及自由能景观计算:结合元动力学等增强采样技术和自由能计算,更有效地探索蛋白质的构象空间,量化不同构象状态的相对稳定性,从而识别和优化与药物结合相关的亚稳态构象。
通过上述协同工作,AlphaFold2-RAVE能够生成多样化的构象系综,识别并富集在无偏MD模拟中处于亚稳态的“holo-like structures”。例如,对于DDR1激酶,AlphaFold2-RAVE成功生成并识别出与晶体结构相似的DFG-out构象,使得II型抑制剂(如伊马替尼和帕纳替尼)能够成功对接,配体RMSD值小于2 Å。该方法在Abl1激酶上也展现了可转移性,通过DDR1的DFG-out模板预测了Abl1的“holo-like structures”,并实现了对II型抑制剂的有效对接。此外,该协议在处理MEK抑制剂时也展现出显著优势,因为它能够识别与抑制剂结合所需的DVF构象,而AlphaFold2通常难以预测到该构象。这表明AlphaFold2-RAVE在构象选择性药物设计中具有巨大应用潜力,特别是对于理解和利用“holo-like structures”的形成和优化药物分子设计具有重要意义。
尽管AlphaFold2-RAVE显著增强了对蛋白质动态构象的预测能力,但其仍存在局限性。该方法的计算成本相对较高,且仍需进一步验证其在更广泛的蛋白质系统中的普适性。即使是衍生工具,在构象覆盖度和准确性方面也可能存在局限性,因此未来发展仍需进一步结合分子动力学模拟或其他增强采样方法。相较于其他动态构象预测方法,AlphaFold2-RAVE的优势在于结合了深度学习的结构预测能力和传统物理模拟的动态探索能力,但其在处理极端构象转换或罕见亚稳态构象时的效率和准确性仍需持续优化与验证。
3.3 特异性与结合亲和力预测的挑战与多尺度建模
AlphaFold2 在高精度蛋白质结构预测方面取得了显著成就,但其在特异性配体结合位点识别和结合亲和力预测方面仍存在固有局限性,这直接影响了药物发现过程中的效率与成功率 。这些局限性主要源于 AlphaFold2 的训练数据以单体蛋白质结构为主,缺乏配体结合状态下的动态信息和复合物结构信息,且模型本身不包含直接的结合亲和力预测模块 。
具体而言,AlphaFold2 主要预测单一稳定构象,这限制了其在处理蛋白质构象柔性方面的能力 。对于需要特定构象(如 II 型抑制剂)才能有效结合的靶点,例如激酶的 DFG-out 构象,AlphaFold2 直接生成的结构往往难以与伊马替尼和帕纳替尼等 II 型抑制剂有效对接,导致较高的假阴性率和较低的对接精度 。这种静态预测的不足使得 AlphaFold2 难以捕捉配体结合诱导的构象变化,从而影响了其在基于结构的虚拟筛选和药物优化中的应用,尤其是在需要考虑蛋白质动态性和配体诱导构象变化的药物设计场景中 。
为了弥补这些不足,AlphaFold2 提供的静态结构应被视为基础,需结合其他计算方法以精确描述蛋白质-配体相互作用 。这些补充方法包括分子动力学(MD)模拟、自由能计算(如自由能微扰 FEP)和量子化学计算等多尺度建模策略 。例如,AlphaFold2-RAVE 的开发尝试通过生成蛋白质动态构象系综来间接解决构象柔性问题,但其仍需依赖下游的分子对接和自由能计算工具来评估结合亲和力 。在实际应用中,计算成本与计算精度之间存在权衡,更高精度的自由能计算通常伴随着显著的计算资源消耗,因此在药物发现流程中需要根据具体需求选择合适的计算方法。
未来的研究应着力于开发结合 AlphaFold2 结构的“特定 AI 模型”,以期实现更高效和精确的结合亲和力预测 。例如,可以探索结合几何深度学习(Geometric Deep Learning)或知识图谱嵌入(Knowledge Graph Embedding)来学习蛋白质-配体相互作用的复杂模式,并直接预测结合亲和力。此外,通过集成基于物理的模拟(如分子动力学模拟、自由能微扰 FEP)与 AI 模型,建立混合模型,能够兼顾计算效率与物理精确性。为支持这些新模型的开发与验证,构建大规模、高质量的“蛋白质-配体复合物结构与结合亲和力数据库”至关重要。这需要结合高通量计算模拟与实验验证,并考虑“多构象”和“变构效应”对结合亲和力的影响,以更全面地捕捉蛋白质-配体相互作用的复杂性 。
3.4 计算资源、数据偏差与模型泛化挑战
AlphaFold2模型在蛋白质结构预测领域取得了显著突破,但其大规模应用,特别是在药物发现研究中,面临着巨大的计算资源需求和数据偏差挑战。AlphaFold2的运行需要高性能计算集群和大量的图形处理器(GPU)算力,尤其是在处理大型蛋白质或复杂系统时,计算成本显著增加,例如AlphaFold2-RAVE协议的应用会进一步推高成本 。这对于中小型研究机构或资源有限的实验室构成了显著的障碍 。
除了计算成本,AlphaFold2的预测性能还受到其训练数据(主要来源于PDB数据库)潜在偏差的影响。这种偏差导致模型在处理特定靶点或蛋白家族(如膜蛋白、柔性区域)时,尤其是在处理高度保守蛋白质时,生成多样化构象的能力受限 。例如,对于Abl1和Src激酶等高度保守的蛋白质,AlphaFold2的Reduced MSA (rMSA)协议难以有效初始化并生成经典的DFG-out构象,这表明训练数据中可能存在的偏差以及模型在处理高度保守序列时泛化能力不足 。这种局限性影响了AlphaFold2在特定蛋白质类型上预测多样化构象的普适性,进而限制了其在某些特定药物靶点上的应用效果,包括在探索非典型结合位点或变构位点时的表现 。此外,对于缺少大量同源序列的蛋白质,AlphaFold2的预测精度也会受到影响 。这种偏差还反映出AlphaFold2在预测蛋白质动态性方面的泛化能力存在局限性,尤其是在需要捕获多个亚稳态构象以进行药物设计时 。
为解决这些挑战,可以提出多方面策略。首先,针对数据偏差问题,可以探索结合非结构化数据源、数据增强、迁移学习或利用非PDB来源数据进行训练。例如,通过引入基于AlphaFold模板的协议(AlphaFold template protocol)可以克服初始化问题,从而为高度保守的激酶生成更多样化的构象 。其次,为扩大训练数据集的规模和多样性,同时保护数据隐私,可以开发“联邦学习(Federated Learning)”框架,允许多个研究机构在不共享原始敏感数据的情况下共同训练AlphaFold2或其衍生模型 。此外,“迁移学习(Transfer Learning)”和“少样本学习(Few-Shot Learning)”技术能够有效应对特定蛋白类型或罕见疾病靶点数据稀疏的问题,从而提高模型的泛化能力。
从技术生态系统层面来看,鼓励开源硬件和软件工具的开发将有助于降低计算门槛,使更多研究者能够利用AlphaFold2的强大功能。最后,建立一个“全球蛋白质结构预测与药物发现数据联盟”至关重要。该联盟可以推动标准化数据格式和共享协议,激励研究者共享训练数据和模型权重,从而系统性地解决数据偏差问题,共同加速药物发现的进程 。
3.5 AI模型透明度、可解释性与实验验证的挑战
尽管AlphaFold2 (AF2) 及其结合 AI 模型在加速药物发现方面展现出巨大潜力,但当前研究中普遍存在的模型透明度、可解释性以及实验验证不足的问题,严重限制了这些技术的普适性、可信度及其在临床转化中的应用。当前研究(如 )普遍未能详细阐述所用 AI 模型(如生成模型或对接算法)的具体算法、架构和训练细节,这构成了 AI 模型“黑箱”特性的核心问题。这种不透明性阻碍了研究人员深入理解 AI 模型如何做出分子设计决策,从而削弱了其在不同靶点或疾病中推广应用的潜力,并使得模型的调试和改进变得困难 。例如,AlphaFold2-RAVE 协议虽然尝试通过潜在空间分析 (SPIB) 来揭示构象状态,但这仍主要停留在模型内部的解释层面,对模型内部工作机制的解释相对简略,限制了进一步优化的可能性 。
为应对 AI 模型透明度与可解释性的挑战,未来研究应致力于开发可解释性 AI (XAI) 方法,以揭示 AI 在分子生成过程中关注的蛋白质结构区域和分子特征。具体而言,可以利用注意力机制、特征归因方法(如局部可解释模型不可知解释,LIME;或 SHAP 值)来可视化 AF2 或结合 AI 模型在预测过程中关注的蛋白质区域或分子特征。此外,引入因果推断模型在药物发现中的应用,旨在揭示特定结构特征与药效之间的因果关系,而非仅仅是统计相关性。为了提升研究的可复现性和透明度,研究人员在发表模型时应提供详细的模型架构、训练数据、训练过程、评估指标以及可复现的代码。同时,开发交互式可视化工具,允许实验科学家和药物化学家直观地探索 AI 模型的预测结果和解释,从而增强对 AI 模型的信任度和采纳率 。
除了 AI 模型透明度问题,当前研究在实验验证方面也存在细节不足的问题,这严重影响了结果的普适性和临床转化潜力。多项研究在提及 AI 设计的分子在实验中显示出积极疗效时,普遍缺乏关于实验条件、细胞系/动物模型选择依据、关键药效学/药代动力学 (PK/PD) 参数以及药物安全性评估(如细胞毒性、脱靶效应)的详细信息 。这种信息缺失导致结果的外部效度和可重复性受到影响,限制了其在真实世界应用中的可靠性。此外,现有研究普遍对 AF2 预测结构在药物设计中具体影响的量化分析不足,缺乏严格的对照研究来理解 AF2 的真正贡献。例如,研究未具体量化 AF2 结构精度对后续 AI 设计分子结合亲和力或特异性的具体贡献,也未与实验结构或低精度预测结构进行严格比较,以说明 AF2 结构带来的显著优势 。
为弥补实验验证的不足,未来研究应建立更完善的计算-实验集成平台,确保计算预测结果能得到详尽、多维度的实验验证。这包括明确指出所使用的实验模型与人类疾病的相关性,并提供详细的剂量-反应曲线、药物安全性评估以及 PK/PD 数据。应开展系统比较研究,量化使用 AF2 预测结构、实验解析结构或传统同源建模结构作为输入时,AI 驱动的药物设计在效率、设计质量和实验验证成功率上的差异。这可通过计算结合自由能(如 MM/GBSA、Free Energy Perturbation)和体外结合实验(如 SPR、ITC)来量化不同结构输入对药物设计结果的影响。建立“计算-实验闭环反馈平台”,实现计算预测、高通量合成、自动化实验验证(如 SPR、ITC、细胞活性筛选)和数据反馈到模型迭代的无缝衔接,同时引入贝叶斯优化 (Bayesian Optimization) 或强化学习 (Reinforcement Learning) 来指导实验设计和数据采集,以最大化信息增益。建议所有与计算预测相关的药物发现研究都应强制提供详尽的实验验证方案、完整的原始数据和透明的统计分析。在报告 AF2 的贡献时,应进行“对照实验”,例如比较使用 AF2 预测结构、实验结构和同源建模结构在药物设计中产生的差异,并量化这些差异对下游实验结果的影响,如通过比较命中率、结合亲和力或细胞活性等指标 。
4. AlphaFold2重塑药物发现范式:前瞻性研究与跨学科融合
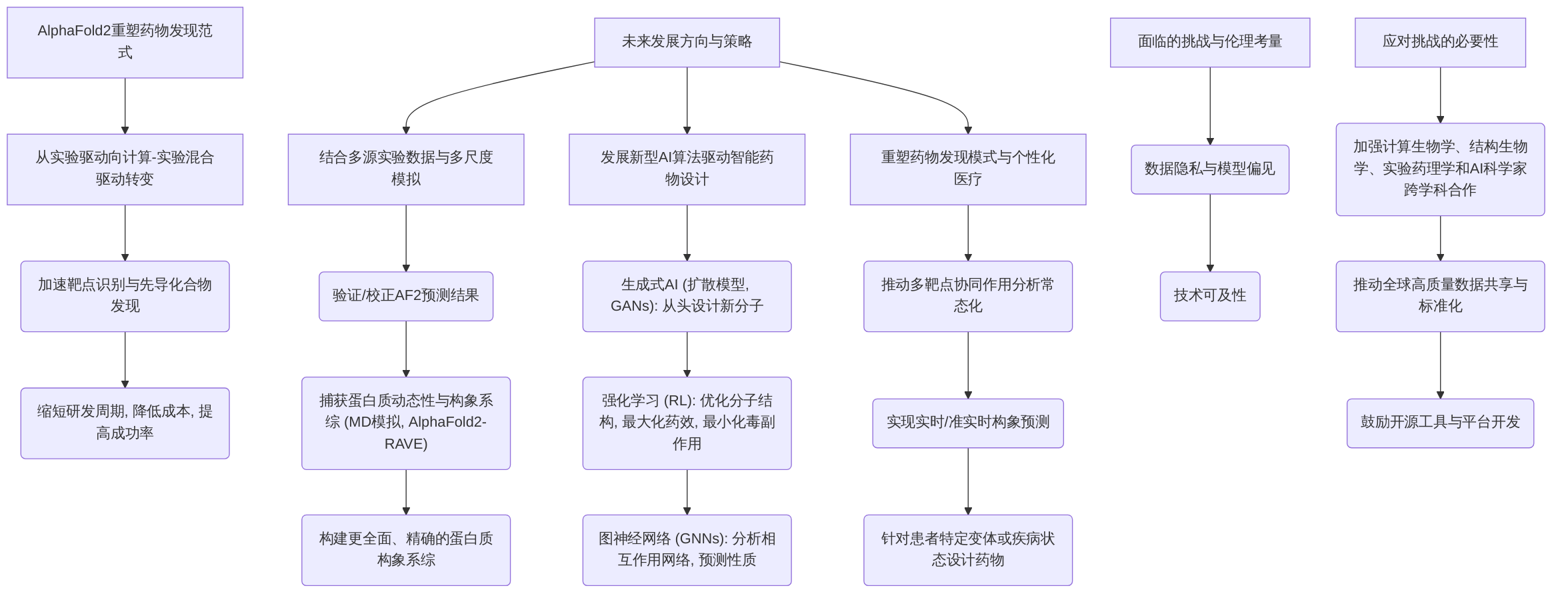
AlphaFold2(AF2)的问世,作为蛋白质结构预测领域的里程碑式突破,正在深刻且持续地重塑小分子药物发现的传统范式 。这种变革不仅体现在加速靶点识别和先导化合物发现等早期阶段,更在于推动药物研发流程从传统的实验驱动向计算与实验紧密结合的混合驱动模式转变,并有望显著缩短研发周期、降低成本、提高新药研发的成功率 。
本章将系统性地展望AlphaFold2引领下的药物发现未来图景,强调如何通过结合多源实验数据与多尺度模拟、发展新型人工智能(AI)算法、以及促进跨学科协同,来克服现有挑战并推动药物发现范式的根本性转变。尽管AlphaFold2在预测蛋白质静态结构方面展现出前所未有的精度,但蛋白质在生理条件下的动态性、构象柔性以及与小分子配体的复杂相互作用,仍是其独立应用时的主要局限。因此,未来的药物发现将更加依赖于AF2的静态预测能力与多维度信息的深度融合,包括长时程分子动力学(MD)模拟、多样化的实验数据以及创新的AI算法,以期全面理解蛋白质构象系综,从而实现更精准的药物设计,特别是构象选择性药物的开发 。
具体而言,本章将首先探讨如何通过整合多源实验数据和多尺度分子模拟,弥补AlphaFold2在蛋白质动态性捕捉方面的不足,构建更全面的蛋白质构象系综。其次,我们将深入分析新型AI算法,如生成式人工智能、强化学习、扩散模型和图神经网络,如何与AlphaFold2深度融合,驱动智能药物设计的创新,实现从头设计具有特定药理性质的分子以及优化药物-靶点相互作用。最后,本章将展望AlphaFold2如何重塑药物发现模式,并为个性化医疗的未来发展奠定基础,同时探讨在这一变革过程中可能面临的伦理考量与挑战,并提出加强跨学科合作与数据共享的必要性,以应对这些挑战。
4.1 结合实验数据与多尺度模拟的协同策略
AlphaFold2(AF2)作为一项变革性的蛋白结构预测技术,在获取蛋白质静态结构方面展现出卓越能力。然而,蛋白质在生理条件下并非静态实体,其动态性和构象柔性对于理解其生物学功能和药物发现至关重要。因此,将AlphaFold2的静态预测能力与多源实验数据及长时程分子动力学(MD)模拟相结合,是克服AF2局限性、提供更全面准确蛋白质结构与动态信息以提升药物发现成功率和作用机制理解的关键策略 。
协同策略的核心在于整合多模态数据,以捕获蛋白质的构象系综。传统的X射线晶体学、冷冻电镜(Cryo-EM)和核磁共振(NMR)等实验技术,结合新型实验方法如小角X射线散射(SAXS)、荧光共振能量转移(FRET)以及稳定膜蛋白类似物(SMPAs),能够提供丰富的蛋白质结构信息和动态特征 。例如,SMPAs技术通过稳定膜蛋白的天然构象,使其更易于结晶并进行高精度结构解析,尤其弥补了AlphaFold2在膜蛋白预测方面的不足 。这些实验数据可用于验证、校正AlphaFold2的预测结果,或作为其训练数据的一部分,从而提高预测能力 。
为了弥补AlphaFold2在捕捉动态性方面的固有局限,多尺度分子模拟,特别是长时程分子动力学(MD)模拟,被广泛引入。MD模拟能够模拟蛋白质在生理环境中的动态行为,探索其构象变化和亚稳态构象,这对于理解药物结合过程至关重要 。AlphaFold2-RAVE协议是这种协同策略的典型范例,它结合了AlphaFold2的结构预测与无偏分子动力学模拟,通过SPIB(State Predictive Information Bottleneck)模型学习反应坐标并进行增强采样,从而计算自由能景观 。该协议通过识别和筛选AlphaFold2生成的结构系综中的“holo-like structures”,即药物结合所需的亚稳态构象,克服了单一静态预测的局限性 。例如,通过伞形采样计算DDR1激酶的自由能分布,并成功识别出具有较低自由能的经典DFG-out构象,从而实现II型抑制剂的精确对接 。这种方法还通过调整多序列比对(MSA)深度和均方根偏差(RMSD)截止值来过滤非物理结构,确保生成构象的质量与多样性 。
展望未来,利用扩散模型(diffusion models)或变分推断(variational inference)从这些多模态数据中学习蛋白质的复杂构象空间,并生成动态的、生理相关的构象轨迹,将是实现构象选择性药物精确设计的关键方向 。通过深度学习方法整合来自不同实验技术和计算模拟的数据,有望构建更加完整和精确的蛋白质构象系综,从而为基于构象选择性的药物设计提供更可靠的基础,显著提高药物发现的成功率和对作用机制的深入理解。
4.2 新型AI算法驱动的智能药物设计
展望未来,AlphaFold2在蛋白质结构预测领域的突破性进展将与一系列新型人工智能算法深度融合,共同驱动智能药物设计迈向新纪元。这种融合旨在弥补AlphaFold2在捕捉蛋白质动态性、精确预测结合亲和力等方面的现有局限性,并最终实现更高效、更智能的药物发现流程。
新型AI算法与AlphaFold2的融合及其应用
生成式人工智能(Generative AI)是未来药物设计领域的核心驱动力之一。它能够基于AlphaFold2提供的高精度蛋白质结构信息,从零开始设计具有特定药理性质(如靶点选择性、吸收、分布、代谢、排泄性质)的新型小分子。具体而言,扩散模型(Diffusion Models)和生成对抗网络(GANs)能够用于生成具有特定结合位点亲和力或构象选择性的分子 。图神经网络(Graph Neural Networks, GNNs)则在分析蛋白质-配体相互作用网络、处理分子结构数据以及预测分子性质和药物-靶点相互作用方面展现出强大潜力 。
强化学习(Reinforcement Learning, RL)的引入将进一步优化药物设计流程。通过迭代学习和反馈机制,强化学习可以逐步改进分子结构,以最大化药效并最小化毒副作用 。在构象搜索和相互作用优化方面,强化学习能够高效地探索药物分子在蛋白质结合口袋中的构象和相互作用空间,从而优化其结合模式 。AlphaFold2-RAVE协议就是人工智能算法在蛋白质结构预测与药物设计领域深度融合的实例,它利用深度学习模型(如SPIB模型)从无偏分子动力学(MD)模拟中学习反应坐标,并通过增强采样技术(如伞形采样)探索蛋白质的构象空间,这不仅增强了AlphaFold2预测蛋白质动态性的能力,也为未来集成更多新型人工智能算法奠定了基础 。
未来发展方向与跨学科合作
AlphaFold2在药物发现领域的未来发展,还将包括其与分子动力学模拟、量子化学计算等新兴技术的结合。这些技术的结合将提供更深入的分子层面理解,从而指导更精确的药物设计。例如,分子动力学模拟可以揭示蛋白质的动态行为和配体结合过程中的构象变化,弥补AlphaFold2在静态结构预测上的不足。
推动上述前沿研究进展的关键在于加强计算生物学、实验生物学和人工智能科学家之间的跨学科合作。这种紧密的协作模式将确保理论模型与实验验证之间的有效衔接,从而加速从靶点识别到先导化合物发现的无缝衔接和全自动化流程 。例如,计算化学家、生物学家和人工智能工程师的紧密协作,是推动这些技术进步的关键 。
综上所述,AlphaFold2与新型人工智能算法的深度融合,如生成式人工智能、强化学习、扩散模型和图神经网络,将共同驱动智能药物设计的发展,实现从头设计具有特定性质的新分子、优化先导化合物,并精准预测药物-靶点相互作用,从而加速药物研发的整体进程 。
4.3 重塑药物发现模式与个性化医疗的未来
AlphaFold2及其衍生的结构预测技术,例如AlphaFold2-RAVE,正在深刻重塑小分子药物发现的范式,推动其从传统的实验驱动模式向计算与实验紧密结合的混合驱动模式转变 。这种转变有望显著缩短药物研发周期,降低研发成本,并提高新药研发的成功率 。
AlphaFold2通过提供高精度的蛋白质结构信息,极大地加速了构象系综的下游应用,例如指导高通量筛选,从而实现更快、更精准的命中化合物识别 。特别是在膜蛋白药物发现领域,SMPAs(稳定膜蛋白类似物)等技术的突破,结合AlphaFold2的结构预测能力,使得约60%已上市药物靶向的膜蛋白能够更容易地获得精确三维结构,进而加速神经退行性疾病和癌症等重要疾病领域的药物研发进程 。AlphaFold2-RAVE协议进一步克服了AlphaFold2在预测动态构象方面的局限性,显著提升了构象选择性药物发现的效率和成功率,尤其在II型激酶抑制剂的发现中展现出巨大潜力,为个性化医疗奠定了基础 。这意味着未来药物研发将能够更好地应对靶点柔性和构象异质性,加速发现具有更高特异性和更少脱靶效应的药物,并能够推广到G蛋白偶联受体(GPCRs)或酶的变构位点等需要考虑蛋白质动态构象的靶点 。
未来药物发现应更加注重计算预测与高通量实验验证之间的紧密耦合,形成一个闭环的“设计-合成-测试-分析”(D-S-T-A)循环。AlphaFold2的引入将推动多靶点协同作用分析的常态化,即同时设计能够作用于多个疾病相关靶点的药物,以提高疗效并减少耐药性 。实时或准实时构象预测的新兴方向将使研究人员能够更好地理解蛋白质在动态环境中的行为,从而设计出更具生理相关性的药物,并为个性化医疗提供基础,允许针对患者特定蛋白质变体或疾病状态下的构象进行药物设计 。
高效利用AlphaFold预测的复杂构象系综信息进行大规模药物筛选和优化是未来的关键方向。这包括开发新的虚拟筛选方法,能够有效地对蛋白质的多个动态构象进行对接和评分。例如,将AI驱动的分子生成与构象系综进行协同,直接生成与特定构象系综匹配的高亲和力配体,并开发高通量计算工具来快速评估这些配体对不同构象的特异性。此外,提升模型的泛化能力和可解释性,以增强模型对未知蛋白质家族或疾病领域的预测效力,是亟待解决的挑战。
这种范式转变可能带来的伦理考量和挑战不容忽视,例如数据隐私、模型偏见和技术可及性。为了应对这些挑战,未来药物发现领域应加强计算生物学、结构生物学、实验药理学和AI科学家之间更紧密的跨学科合作,推动全球范围内高质量的蛋白质结构、相互作用和药物发现数据共享,以克服数据偏差和稀疏性问题,为训练更强大、更通用的AI模型提供基础。鼓励开发标准化数据格式和开源工具,降低研究门槛 。
5. 结论
AlphaFold2的出现标志着蛋白质结构预测领域的一项里程碑式突破,其在加速靶点结构获取、赋能小分子设计与优化、以及提升药物筛选效率方面展现出卓越的潜力,无疑重塑了结构引导药物发现的传统范式 。通过提供高精度的蛋白质结构模型,AlphaFold2极大地降低了药物研发的时间和成本,并有望提高研发成功率 。
然而,尽管AlphaFold2取得了显著成就,其在应用于小分子药物发现过程中仍面临多重挑战。首先,在捕捉蛋白质动态性和柔性方面存在局限性,AlphaFold2通常预测单一稳定构象,难以有效识别和利用蛋白质的亚稳态构象,这对于构象选择性药物设计构成了显著障碍 。其次,AlphaFold2在膜蛋白结构预测方面仍有待提升,尤其是在处理这类复杂体系时可能存在的局限性,这限制了其在靶向膜蛋白药物发现中的应用 。此外,结合亲和力预测的精确性、高计算资源需求、潜在的数据偏差以及AI模型的可解释性与透明度不足等问题也持续构成挑战 。现有研究还指出,实验验证的完整性不足以及AlphaFold结构对药物发现贡献的量化不足,限制了对其全面评估和推广应用 。
| 挑战类别 | 具体挑战点 | 应对策略 | 预期成果/未来展望 |
|---|---|---|---|
| 蛋白质动态性与柔性 | 预测单一稳定构象,难捕获亚稳态构象;构象选择性药物设计障碍 | AlphaFold2-RAVE协议 (结合MD模拟、先进采样、机器学习);优化MD模拟效率;集成更先进AI算法 | 有效识别和利用亚稳态构象;提升构象选择性药物发现效率;实现全流程自动化与智能化 |
| 膜蛋白结构预测 | 膜蛋白训练数据少,预测精度有限;难处理复杂体系 | 稳定膜蛋白类似物 (SMPAs) 技术;开发针对多链复合物、PPI、PTMs的AI模型 | 提供关键实验结构信息,与AF2互补;加速膜蛋白靶向药物发现;应对更广泛疾病挑战 |
| 结合亲和力预测 | 训练数据缺乏配体结合动态信息;模型无直接亲和力预测模块 | 结合AI模型进行结合亲和力预测;集成基于物理的模拟 (MD, FEP);构建高质量蛋白质-配体复合物数据库 | 实现更高效、精确的结合亲和力预测;兼顾计算效率与物理精确性 |
| 计算资源与数据 | 高计算资源需求;训练数据偏差;泛化能力不足 | 联邦学习、迁移学习、少样本学习;开源硬件与软件工具开发;建立全球数据联盟 | 降低计算门槛;克服数据偏差;提高模型泛化能力;加速药物发现进程 |
| 模型透明度与验证 | AI模型“黑箱”;可解释性不足;实验验证细节缺乏;AF2贡献量化不足 | 可解释性AI (XAI) 方法;因果推断模型;提供详细模型信息与代码;建立计算-实验闭环反馈平台;开展对照研究 | 揭示模型决策过程;增强可信度与普适性;确保计算预测结果能得到详尽、多维度的实验验证 |
| 范式重塑与个性化医疗 | - | 推动药物发现从试错法向计算-实验混合驱动转变;多靶点协同作用分析;实时构象预测;应对伦理、社会和技术可及性挑战 | 更高效、低成本、个性化的药物研发;加速精准医疗进程;实现“智能设计-快速合成-高效测试-智能分析”循环 |
为应对上述挑战,未来研究应聚焦于多方面创新。针对蛋白质动态性问题,AlphaFold2-RAVE等协议的提出,通过结合AlphaFold2的预测能力与分子动力学(MD)模拟、先进采样技术以及机器学习方法,已被证明能够有效识别和利用蛋白质的亚稳态构象,尤其在构象选择性药物发现中展现出识别“holo-like structures”的关键能力 。未来的方向包括提升AlphaFold2在处理高保守蛋白时的构象多样性预测能力,优化MD模拟效率,并整合更先进的AI算法以实现全流程自动化和智能化 。针对膜蛋白挑战,稳定膜蛋白类似物(SMPAs)等技术通过稳定膜蛋白的天然构象,促进高分辨率结构解析,与AlphaFold2形成互补,为结构引导药物设计提供了关键实验结构信息 。总而言之,将AlphaFold2与实验数据、多尺度模拟、新型AI算法(如生成式AI、强化学习、图神经网络)深度融合,有望克服现有局限性,实现更精准的构象系综分析、多靶点协同作用分析以及个性化药物设计 。
AlphaFold2及其后续技术正推动药物发现从传统的试错法向计算-实验混合驱动模式的根本性转变,从而实现更高效、低成本、个性化的药物研发 。这不仅加速了结构引导药物发现的进程,更预示着精准医疗的广阔前景。未来的研究应进一步探索如何实现更高效、更具可解释性的AI模型,以支持“从头设计”和“精准优化”的药物发现策略;如何真正将计算与实验形成无缝的反馈闭环,实现“智能设计-快速合成-高效测试-智能分析”(D-S-T-A)的研发循环;如何将AlphaFold2的优势拓展至更复杂的生物系统和多靶点药物发现,例如蛋白质复合物,从而应对更广泛的疾病挑战;以及如何有效应对随之而来的伦理、社会和技术可及性挑战,确保AI技术在药物发现领域的公平、负责任应用。
References
AlphaFold2 can be an effective tool in structure-based drug discovery - News-Medical.net https://www.news-medical.net/news/20240517/AlphaFold2-can-be-an-effective-tool-in-structure-based-drug-discovery.aspx
Integrating AlphaFold into the Drug Discovery Process - Creative Biostructure https://www.creative-biostructure.com/integrating-alphafold-drug-discovery.htm
Empowering AlphaFold2 for protein conformation selective drug discovery with AlphaFold2-RAVE - eLife https://elifesciences.org/reviewed-preprints/99702
The Revolutionary Impact of AlphaFold on Drug Discovery: Decoding the Mystery of Protein Folding - Lindus Health https://www.lindushealth.com/blog/the-revolutionary-impact-of-alphafold-on-drug-discovery-decoding-the-mystery-of-protein-folding
Empowering AlphaFold2 for protein conformation selective drug discovery with AlphaFold2-RAVE - PubMed https://pubmed.ncbi.nlm.nih.gov/39240197/
AlphaFold2: Transforming Structure-Based Drug Discovery | The Lifesciences Magazine https://thelifesciencesmagazine.com/alphafold2-structure-drug-discovery/
AlphaFold2 structures guide prospective ligand discovery - PubMed https://pubmed.ncbi.nlm.nih.gov/38753765/
Empowering AlphaFold2 for protein conformation selective drug discovery with AlphaFold2-RAVE - arXiv https://arxiv.org/html/2404.07102v2
New study uses AlphaFold and AI to accelerate design of novel drug for liver cancer https://www.artsci.utoronto.ca/news/new-study-uses-alphafold-and-ai-accelerate-design-novel-drug-liver-cancer
Membrane protein analogues could accelerate drug discovery - News - EPFL https://actu.epfl.ch/news/membrane-protein-analogues-could-accelerate-drug-d/
